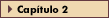 FIGURA 2-3 FIGURA 2-3
Modelo para a interação dos receptores de insulina (IR – insulin receptors) e moléculas de substrato do receptor de insulina (IRS – insulin receptor substrate). As proteínas IRS foram identificadas como proteínas fosforiladas em tirosina nas células tratadas com insulina [26–28]. Além da insulina, outros agentes como os fatores de crescimento semelhantes à insulina e interleucinas estimulam a fosforilação de proteína IRS [20]. Após ativação da IR quinase, o domínio de ligação à fosfotirosina (PTB – phosphotyrosine-binding) do IRS torna-se intimamente associado à região justamembranar do IR. Esta interação exige fosforilação da tirosina 972 no IR. Além disto, é possível que o domínio de homologia da plecstrina (PH) estabilize esta interação. O domínio PH está localizado na extremidade aminoterminal da proteína IRS, que poderia promover a interação com a bicamada membranar. Muitos domínios PH se ligam a fosfolípides. Além disto, existe pelo menos um domínio adicional que interage com o IR, chamado domínio de ligação à alça regulatória da quinase (KRLB – kinase regulatory loop-binding). Sua interação com o IR exige fosforilação de resíduos de tirosina no domínio catalítico do receptor (tirosinas 1158, 1162 e 1163). Por estar presente apenas em Irs-2, o KRLB poderia determinar especificidade da interação entre o IR e seus substratos [29,30]. Experimentos de ablação gênica em camundongos indicam que as várias proteínas IRS têm funções singulares. Camundongos nos quais Irs-1 está ausente apresentam retardo de crescimento e são discretamente resistentes à insulina, sugerindo que o Irs-1 desempenha um importante papel na função do receptor tanto da insulina quanto do fator-1 de crescimento semelhante à insulina; camundongos com ausência de Irs-2 desenvolvem resistência à insulina e apresentam desenvolvimento comprometido de células b, sugerindo que o Irs-2 é importante para regulação metabólica em resposta à insulina [4,21].
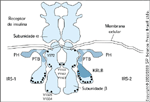
 FIGURA 2-4 FIGURA 2-4
Estrutura das proteínas IRS. As proteínas IRS não têm atividades catalíticas intrínsecas, mas são compostas de vários domínios de interação e de motifs de fosforilação. Ocorrem pelo menos três proteínas IRS em camundongos e pessoas, que incluem o IRS1 e o IRS2 que são amplamente expressos, enquanto o IRS4 está limitado ao timo, cérebro, rim e, possivelmente, células b pancreáticas [31]. Os roedores também expressam o Irs3, que está basicamente restrito ao tecido adiposo e exige uma atividade semelhante ao do Irs1; entretanto, é possível que este curto ortolog não ocorra em seres humanos [32]. A análise filogenética revela uma íntima relação evolutiva entre IRS1/Irs1 e IRS2/Irs2 de pessoas/camundongos, que podem ter divergido a partir de IRS4/Irs4. A proteína IRS de Drosophila, Chico, está fracamente relacionada com seus ortologs mamíferos já que contém poucos sítios de fosforilação de tirosina terminais. Finalmente, a análise de seqüência do genoma humano revela pelo menos 2 proteínas IRS putativas reconhecidas por domínios adjacentes de homologia da plecstrina (PH) e de ligação à fosfotirosina (PTB); entretanto, elas contêm caudas COOH bem curtas com poucos sítios de fosforilação da tirosina e, portanto, sua função permanece desconhecida (veja a Fig. 2-2).Todas as proteínas IRS são caracterizadas pela presença de um domínio PH NH2-terminal adjacente a um domínio PTB, seguidos por uma cauda COOH-terminal de comprimento variável que contém inúmeros sítios de fosforilação de tirosina e de serina. Os domínios PH e PTB medeiam interações específicas com as quinases de receptor da insulina e do fator-1 de crescimento semelhante à insulina (IGF-1) [33,34]. O domínio PTB se liga a motifs NPXY fosforilados nos receptores da insulina, do IGF-1 ou da interleucina-4; entretanto, outros receptores que promovem a fosforilação de tirosina da proteína IRS não contêm motifs NPXY [35]. Os candidatos a parceiros de ligação ao domínio PH poderiam incluir fosfolípides, peptídios ácidos ou proteínas específicas como PHIP [36,37]. Entre as proteínas que comprovadamente se ligam ao IRS1, a mais proeminente é a subunidade p85 da fosfatidilinositol 3-quinase (PI 3-K), cuja atividade enzimática acredita-se que seja crucial na estimulação de muitas ações biológicas da insulina.
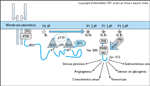 FIGURA 2-5 FIGURA 2-5
Ocorre fosforilação de tirosina em vários motifs de seqüência de aminoácidos que criam sítios de ligação para os assim chamados motifs SH2 (de src homology 2) [38]. Os motifs SH2 têm 50 a 100 aminoácidos de comprimento e se ligam com alta afinidade a resíduos de fosfotirosina em várias moléculas de sinalização intracelular. Durante a estimulação insulínica, as proteínas de substrato do receptor da insulina (IRS – insulin receptor substrate) fosforiladas em tirosina se ligam aos dois domínios SH2 na subunidade regulatória (p85) da fosfatidilinositol 3-quinase (PI 3-K), resultando na ativação da subunidade catalítica p110. A subunidade p110 catalisa a fosforilação do fosfatidilinositol (PI) na posição D3 do anel de inositol, levando à geração de PI 3-fosfato a partir de PI, de PI 3,4-bisfosfato a partir de PI 4-fosfato e de PI 3,4,5-trisfosfato a partir de PI 4,5-bisfosfato. Acumulam-se as evidências de que os inositídeos fosforilados em D3 agem como mensageiros intracelulares que levam à ativação de quinases dependentes de PI, alterações no tráfego intracelular e estimulação do crescimento [39]. Os produtos da PI 3-quinase interagem com serina quinases citosólicas (PDK, PKB e outras) para gerar um sistema de sinalização ligada à membrana que medeia diversos processos biológicos, inclusive síntese de glicogênio e transporte de glicose. O domínio PH da PKB liga-se ao PI 3-fosfato, mas uma atividade enzimática máxima exige fosforilação da Thr308 e da Ser473 mediada pela PDK [40]. Além de seu papel na regulação metabólica, a PKB promove a inibição da apoptose mediada pelo fator-I de crescimento semelhante à insulina, bem como a ativação da p70S6k quinase com as concomitantes síntese proteica, regulação da entrada no ciclo celular e diferenciação celular [41]. PKB — proteína quinase B; PDK — proteína quinase dependente de fosfoinositídeo.
voltar |